人工智能预测“光谱指纹”,侦查新冠蛋白“作恶”过程
发布时间:2021-09-02
近日,材料科学姑苏实验室G2114项目负责人江俊及其团队结合量子化学计算和人工智能技术,发展了一套可高效模拟蛋白质紫外和红外光谱的计算方案,用于模拟新冠病毒S蛋白与人体hACE2蛋白结合过程中的结构变化。相关研究成果以“AI-based spectroscopic monitoring of real-time interactions between SARS-CoV-2 and human ACE2”为题发表在《美国国家科学院院报》(Proc. Natl. Acad. Sci. U.S.A. 118 (2021) e2025879118)上。

图1:人工智能AI“识别”新冠蛋白质“光谱指纹”
新型冠状病毒(SARS-CoV-2)的大流行对全人类健康构成了巨大威胁。生命科学研究证实新冠病毒的受体是人血管紧张素转换酶2(hACE2),病毒通过表面针刺的S糖蛋白与hACE2结合,识别并入侵人体细胞。因此,探测新冠病毒S蛋白识别hACE2并与之结合这一系列过程的结构演变,对开发相关药物和疫苗尤为关键。
光谱虽然能测量分子动态演变,但实验光谱信号必须要经过理论模拟的解读和比对,才能揭示对应的结构信息。蛋白质分子含有成千上万原子,环境涨落对结构的影响显著,结构自由度极大,造成用量子化学方法计算蛋白质分子光谱极其昂贵,制约了蛋白质实验光谱的高效解析,最终阻碍了用谱学来探测蛋白质的动态结构。因此,用光谱技术揭示新冠蛋白与人体蛋白复合物的动态结构信息,需要一套可高效模拟蛋白质分子光谱的计算化学方案。
为了解决蛋白质光谱快速精确模拟的难题,研究人员结合了量子化学计算和人工智能(AI)技术,发展了一套可高效模拟蛋白质紫外和红外光谱的计算方案,前期成果于2019年和2020年,分别发表在国际知名期刊《美国国家科学院院报》(Proc. Natl. Acad. Sci. U.S.A. 116 (2019) 11612-11617.)和《美国化学会志》(J. Am. Chem. Soc. 142 (2020) 19071-19077)上。
本项工作为了实现光谱快速模拟,研究人员运用神经网络技术学习光谱模拟所需要的重要物理参数与分子结构特征之间的关联(如图2所示)。
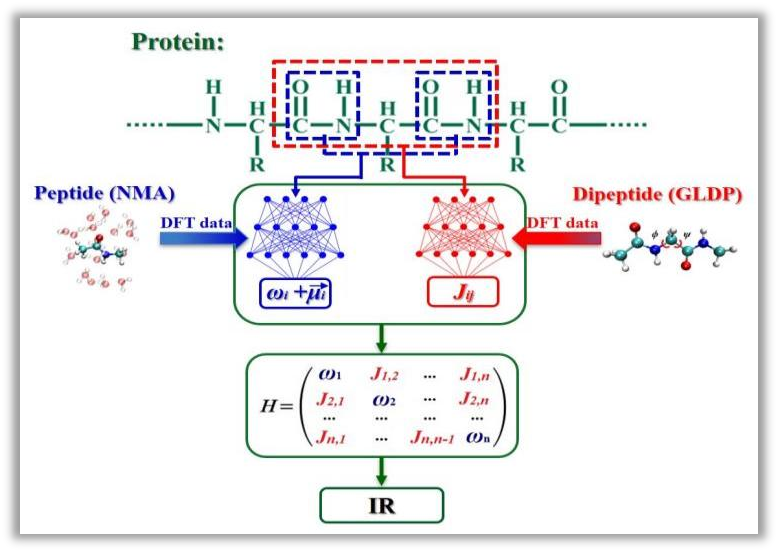
图2:AI模拟生物大分子光谱方案
为了实现光谱快速模拟,研究人员分别以N-甲基乙酰胺分子(NMA)分子和甘氨酸二肽(GLDP)分子为肽键和二肽分子模型,训练了各自的神经网络模型来分别预测肽键振动性质和二肽的振动耦合性质,同时采用偶极-偶极相互作用公式来估算非邻近肽键之间的振动耦合系数,构建了蛋白质体系的振动激子哈密顿量,进而计算得到蛋白质的红外光谱。这套基于AI技术的光谱模拟方案,相对常规的量子化学计算方案运算速度提升了3~4个数量级。
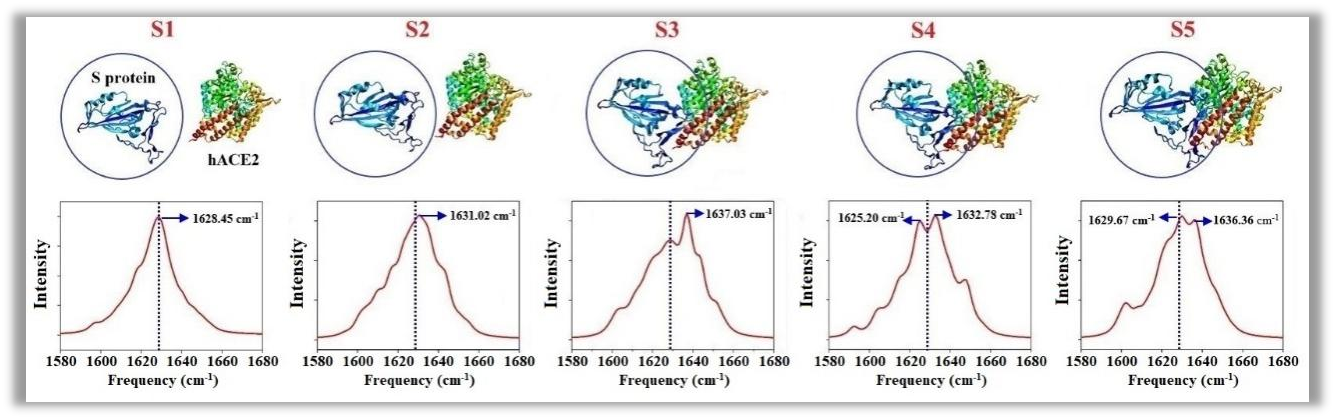
图3:AI模拟新冠S蛋白与hACE2蛋白结合过程的红外光谱变化
基于上述AI光谱模拟方案,研究人员模拟了新冠S蛋白与hACE2蛋白结合过程不同阶段的红外光谱变化,并在此基础上探讨了红外光谱信号变化与二级结构变化之间的关系。如图3所示,蛋白二级结构的不同变化形式,均反映在红外光谱的蓝移或者红移上。例如,S蛋白α-螺旋含量的增加,β-转角的增加和卷曲成分的减少,均会导致红外光谱信号蓝移;而α-螺旋、β-转角的减少,和卷曲成分的增加,导致红外光谱信号红移。
这套AI红外光谱模拟技术,不仅建立了蛋白质二级结构的变化与红外光谱信号变化之间的关联,而且具备快速解析红外光谱的能力。与蛋白质分子红外光谱数据库相结合,AI光谱模拟技术将能快速将实验测量的瞬态红外光谱信息翻译为蛋白质瞬态结构的信息,进而跟踪蛋白质结构变化过程。
本项研究工作表明,基于人工智能技术的光谱模拟方案,能显著加速对蛋白质动态结构的光谱解读,验证了光谱实时跟踪病毒蛋白的结构变化过程的可能性,为发展实验光谱技术揭示新冠病毒入侵人体细胞的过程提供坚实的理论和技术支持。人工智能技术赋能传统量化模拟,将显著促进谱学对生物大分子结构和功能演化的动态跟踪能力。
原文链接:https://www.pnas.org/content/118/26/e2025879118.















 苏公网安备 32059002003194号
苏公网安备 32059002003194号